Procedimientos experimentales
No se utilizaron métodos estadísticos para predeterminar el tamaño de la muestra. Los experimentos no fueron aleatorizados. Los investigadores no estaban cegados a la asignación durante los experimentos y la evaluación de resultados.
Ratones
Se adquirieron ratones 129SV de tipo salvaje en Taconic Biosciences. Todo el trabajo con ratones se realizó cumpliendo con todas las regulaciones éticas relevantes establecidas por el Comité de Cuidado y Uso de Animales Institucional (IACUC) del Hospital de Niños de Boston y bajo protocolos aprobados por el IACUC del Hospital de Niños de Boston. Los ratones se mantuvieron en un entorno controlado de temperatura (22 ± 3 °C) y humedad (35% ~ 70% ± 5%), con acceso libre a comida y agua, siguiendo un horario de luz de 14 horas y oscuridad de 10 horas. Se usaron ratones machos y hembras por igual para todos los experimentos.
Generación y caracterización del modelo de ratón con inversión de todo el locus Vκ
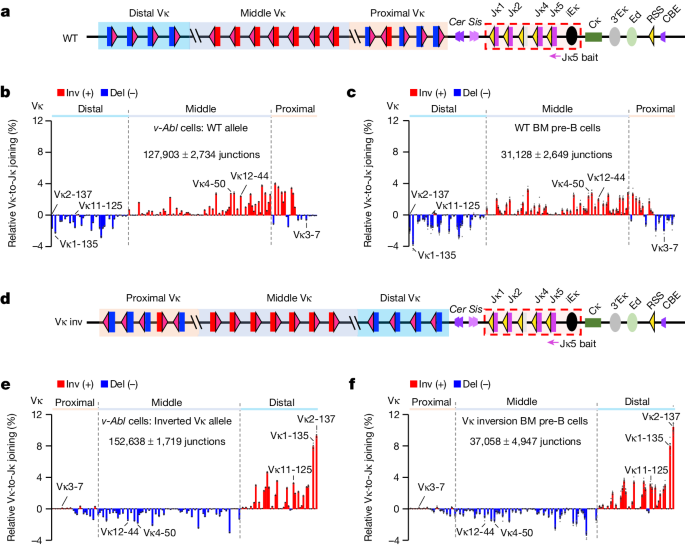
Las modificaciones de la inversión de todo el locus Vκ mediadas por CRISPR-Cas9 se realizaron en un alelo Igk en la línea de células madre embrionarias (ES) TC1. La orientación de las células ES se realizó utilizando sgRNA1 y sgRNA2 como se describió anteriormente. Los clones positivos con inversión del locus Vκ de 3,1 Mb fueron identificados por PCR y confirmados por secuenciación de Sanger. Después de dar negativo para micoplasma, el clon ES con inversión de Vκ se inyectó en blastocistos deficientes en RAG2 para generar quimeras. Los ratones quiméricos se cruzaron con ratones 129SV de tipo salvaje para la transmisión germinal de la inversión dirigida, y se criaron en homocigosis.
Generación del modelo de ratón pre-reorganizado VH7-3 Igh; modelo de ratón Rag2−/−
Los ratones pre-reorganizados VH7-3 Igh heterocigotos o homocigotos (VH7-3wt/re o VH7-3re/re) se generaron mediante células madre pluripotentes inducidas (iPS) y se mantuvieron en el laboratorio de Alt. Para llevar a cabo experimentos 3C-HTGTS con antecedentes deficientes en RAG2, se cruzaron ratones VH7-3wt/re o VH7-3re/re con ratones Rag2−/− para obtener ratones VH7-3wt/re; Rag2−/− o VH7-3re/re; Rag2−/− en el fondo 129SV.
Purificación de precursores de células B de la médula ósea
Para el análisis de RAG en la diana y fuera de la diana, se obtuvieron suspensiones de células individuales de la médula ósea de ratones 129SV machos y hembras de 4 a 6 semanas y ratones con inversión del locus Vκ de Igk y se incubaron en buffer lizante de glóbulos rojos (Sigma-Aldrich, R7757) para eliminar los eritrocitos. Las células B pre-B B220+CD43lowIgM− se aislaron tiñéndolas con anti-B220-APC, anti-CD43-PE y anti-IgM-FITC y purificándolas mediante citometría de flujo por fluorescencia, y las células pre-B primarias purificadas se usaron directamente para HTGTS-V(D)J-seq como se describe.
Para los experimentos 3C-HTGTS, las células pre-B primarias positivas al B220 se purificaron mediante anti-B220 MicroBeads de ratones machos y hembras de 4 a 6 semanas de VH7-3wt/re; Rag2−/− o VH7-3re/re; Rag2−/−. Se agruparon células purificadas de 3 o 4 ratones para cada experimento 3C-HTGTS. Cada ratón fue verificado y confirmado mediante PCR y secuenciación de Sanger antes de varios ensayos.
Generación de la línea celular única Jκ5 v-Abl y sus derivados
Las técnicas de orientación sgRNA-Cas9 descritas para esta sección y todos los párrafos siguientes que describen las modificaciones de la línea v-Abl de v-Abl se realizaron como se describió anteriormente. Todas las líneas celulares de v-Abl no se han probado para la contaminación por micoplasmas.
La línea inicial ‘parental’ de células v-Abl Rag2−/−;Eμ-Bcl2+ en el fondo 129SV se generó previamente. Se introdujeron códigos de barras 1-4 bp aleatorios en un sitio ~85 bp aguas abajo del heptámero RSS Jκ5 y ~40 bp aguas arriba del iniciador señuelo Jκ5 en ambos alelos en esta línea parental, de manera similar al enfoque previamente descrito para modificar JH46. La línea ‘Jκ5-codificada’ v-Abl resultante se dirigió más con sgRNA1 y sgRNA2 para invertir todo el locus Vκ en un alelo y dejar el otro alelo intacto. Por lo tanto, el código de barras específico del alelo Igk permite la separación de las lecturas de secuenciación derivadas del alelo salvaje y el alelo Vκ invertido evaluado con el mismo iniciador señuelo bajo el mismo contexto celular. Esta línea codificada se utilizó para generar los datos en la Figura 1b,e.
Para facilitar más modificaciones en el locus Igk, la línea ‘Jκ5-codificada’ de v-Abl se dirigió con sgRNA1 y sgRNA3 que eliminaron todo el locus Igk en un alelo y dejaron el otro alelo intacto. El código de barras no fue relevante para estudios adicionales basados en esta línea de un solo alelo Igk o sus derivados. La línea de un solo alelo Igk luego se dirigió con otras dos parejas de sgRNAs para eliminar por separado Jκ1 a Jκ4 y el aguas abajo Igk-RS para excluir reorganizaciones secundarias equívocas y mantener la configuración inalterada entre Jκ5 e iEκ. Esta línea se conoce como ‘línea de un solo alelo Jκ5’.
La línea de un solo alelo Jκ5 se modificó aún más con Cas9-sgRNA específicamente diseñado para generar la línea de inversión Jκ5-Vκ (sgRNA8 y sgRNA9), la línea de inversión Jκ5 (sgRNA10 y sgRNA11), la línea de un solo alelo Jκ5-un solo Igh (sgRNA12 y sgRNA13), la línea de un solo Jκ5-PKO (sgRNA2 y sgRNA14), la línea de un solo Jκ5-eliminación de Cer (KO) (sgRNA15 y sgRNA16), la línea de un solo Jκ5-eliminación de Sis (KO) (sgRNA17 y sgRNA18), y la línea de un solo Jκ5-eliminación de CerSis (KO) (sgRNA15 y sgRNA18).
La línea de un solo alelo Jκ1 v-Abl se generó a partir de la línea de un solo alelo Igk mediante la eliminación separada de Jκ2 a Jκ5 y la eliminación aguas abajo Igk-RS.
Todos los clones candidatos con modificaciones génicas deseadas se examinaron mediante PCR y se confirmaron mediante secuenciación de Sanger.
Generación y análisis de líneas celulares WAPL-degron pre-reorganizadas de DJH
Las líneas de v-Abl pre-reorganizadas de DJH en el fondo C57BL/6 se derivaron de la línea v-Abl WAPL-degron previamente descrita. Las secuencias del marco de lectura abierta de Rag1 y Rag2 se clonaron en el vector pMAX-GFP siguiendo el protocolo estándar para generar los plásmidos pMAX-Rag1 y pMAX-Rag2. Estos dos plásmidos (cada uno 2,5 μg) se nucleofectaron en 2,0 x 10^6 células v-Abl WAPL-degron para permitir las reorganizaciones D a JH endógenas mediadas por la expresión transitoria de RAG. Las células que albergan la reorganización DQ52JH4 deseada (línea DJH-WT) se identificaron posteriormente mediante cribado de PCR y verificación mediante secuenciación de Sanger. La línea DJH-inv de v-Abl se generó a partir de la línea DJH-WT utilizando Cas9-sgRNA para apuntar secuencias aguas abajo de JH4 y aguas arriba de DQ52.
Generación de la línea celular híbrida Igh–Igk v-Abl y sus derivados
La línea celular híbrida Igh–Igk se derivó de la línea de un solo alelo Jκ5. En resumen, la línea de un solo alelo Jκ5 se dirigió con sgRNA12 y sgRNA13 para generar la línea de un solo alelo Jκ5-un solo Igh, donde se eliminó el locus Igh completo de un alelo. La línea de un solo alelo Jκ5-un solo Igh se dirigió con sgRNA22 y sgRNA8 para generar una translocación cromosómica equilibrada entre los cromosomas 12 y 6. En la línea de Igh–Igk híbrida resultante, se añadió todo el locus IgkIgh, y el locus VH de IghIgh–Igk híbrida-Vκ, se modificó secuencialmente la línea híbrida Igh–Igk para invertir todo el locus Vκ, mutar los RSS DQ52 y eliminar todos los segmentos D aguas arriba. Para generar la línea Igh–Igk híbrida-Vκ-JκRSS-PKO de la línea Igh–Igk híbrida-Vκ-JκRSS, se utilizaron sgRNA2 y sgRNA14 para eliminar el dominio Vκ proximal.
Para generar la línea Igh–Igk híbrida-D-JH, la línea híbrida Igh–Igk se dirigió con sgRNA27 y sgRNA28 para eliminar IGCR1 y todo el locus Vκ. La línea híbrida Igh–Igk híbrida-D-JH se modificó más para generar la línea Igh–Igk híbrida-D donde se ha eliminado JH1-4.
Todos los clones candidatos con modificaciones génicas deseadas se examinaron mediante PCR y se confirmaron mediante secuenciación de Sanger. Consulte la Figura 4a y las Figuras 5a y 8a de Datos Extendidos para obtener información detallada sobre la estrategia y el procedimiento.
Pintura de cromosomas completos
La pintura de cromosomas completos se realizó en la línea de un solo alelo Jκ5-un solo Igh v-Abl y la línea Igh–Igk híbrida de v-Abl utilizando sondas fluorescentes que cubren todo el cromosoma 6 (Chr6-FITC) y el cromosoma 12 (Chr12-TxRed) según el protocolo estándar. Las células se trataron con colcemid a una concentración final de 0,05 μg/ml durante 3 horas antes de procesarse para la gota metafásica. Las láminas se deshidrataron en una serie de etanol, se desnaturaron a 70 °C durante 1,5 minutos, y se hibridaron a la mezcla de sondas desnaturadas a 37 °C durante 12-16 horas. Luego, las láminas se lavaron, se tiñeron con DAPI y se fotografiaron con un microscopio Olympus BX61. Se utilizó ImageJ (1.53q) para el procesamiento de imágenes.
Experimentos de reemplazo de RSS
Todas las modificaciones de reemplazo de RSS se realizaron mediante Cas9-sgRNA utilizando oligonucleótidos de ADN de cadena simple (ssODN) como plantilla de donante. En resumen, se co-transfectaron 2,5 μg de plásmido Cas9-sgRNA y 5 µl de 10 μM de ssODN en 2,0 x 10^6 células v-Abl. Se llevaron a cabo cribados de PCR secuenciales en clones agrupados y luego en clones individuales, y posteriormente se verificaron mediante secuenciación de Sanger. Específicamente, se utilizaron sgRNA31 y ssODN2 para reemplazar JH1-RSS con Jκ5-RSS en la línea Igh–Igk híbrida-Vκ v-Abl para generar la línea Igh–Igk híbrida-Vκ-JκRSS; sgRNA32 y ssODN3 se usaron para reemplazar Jκ5-RSS con JH1-RSS en la línea de un solo alelo Jκ5-un solo Igh para generar la línea de un solo alelo Jκ5-un solo Igh-JHRSS; sgRNA33 y ssODN4 se utilizaron para reemplazar DQ52 upstream RSS con Vκ12-44-RSS en la línea híbrida Igh–Igk-D