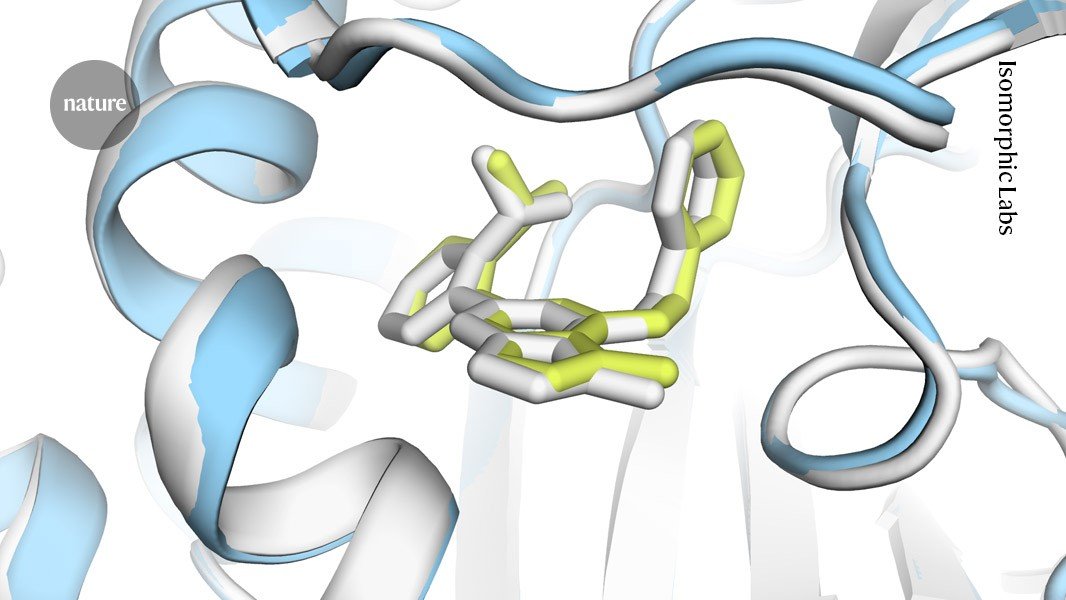
Cuando Google DeepMind mostró AlphaFold3, la última versión de su revolucionaria inteligencia artificial de predicción de la estructura de proteínas en Nature este mes, hubo un problema. A diferencia de una versión anterior, no había código informático que describiera el avance para acompañar al artículo.
La empresa con sede en Londres cambió de opinión días después, prometiendo liberar el código a fin de año. Sin embargo, la omisión ha llevado a investigadores de todo el mundo a desarrollar sus propias versiones de código abierto de AlphaFold3, un modelo de inteligencia artificial que puede predecir la estructura de una proteína, así como la de otras moléculas, incluidos posibles nuevos fármacos. Otros científicos están haciendo todo lo posible para hackear la versión web de AlphaFold3 que DeepMind lanzó para evadir sus limitaciones.
«Sería malo que capacidades tan fundamentales para nuestra capacidad de descubrir medicamentos y otras cosas relevantes para la salud humana terminaran siendo bloqueadas», dice Mohammed AlQuraishi, biólogo computacional en la Universidad de Columbia, en Nueva York. Su equipo ‘OpenFold’ ya ha comenzado a codificar una versión de código abierto de AlphaFold3 que esperan completar este año.
La retención inicial por parte de DeepMind del código de AlphaFold3, así como la publicación del 9 de mayo en Nature, irritaron a muchos científicos. Las políticas de Nature dicen que el código asociado con los estudios debe estar disponible, aunque reconocen que puede haber restricciones.
En un editorial publicado el 22 de mayo, Nature dijo que acogió con satisfacción la conversación que provocó la publicación de AlphaFold3, y busca opiniones de los lectores sobre cómo puede fomentar la apertura en la ciencia. Añadió que sus políticas respaldan la ciencia abierta, pero reconoce que el sector privado financia la mayor parte de la investigación mundial y que muchos descubrimientos que surgen de ese trabajo siguen siendo propietarios. «En Nature pensamos que es importante que las revistas se relacionen con el sector privado y trabajen con sus científicos para que puedan presentar sus investigaciones para revisión por pares y publicación», afirmó la revista. La revista dijo que actualizaría el artículo con el código cuando DeepMind lo publique.
En lugar de código y parámetros obtenidos del entrenamiento de AlphaFold3 llamados pesos del modelo, DeepMind creó un sitio web donde los investigadores pueden acceder a la herramienta. Pero este servidor de AlphaFold3 tiene restricciones: solo se puede utilizar para investigación no comercial, y no es posible obtener estructuras de proteínas unidas a posibles fármacos. El artículo que describe AlphaFold3 también contiene un «pseudocódigo» detallado que describe cómo funciona el modelo.
La disponibilidad de una versión completa de código abierto de AlphaFold3 permitirá a los investigadores comprender mejor cómo funciona el modelo y ampliar sus capacidades. Algunos científicos ya están tratando de hacer esto con el servidor de AlphaFold3. AlQuraishi espera que el impulso para desarrollar versiones de código abierto de AlphaFold3 sirva como una «historia de advertencia» para los académicos sobre los peligros de depender de empresas tecnológicas como DeepMind para desarrollar y distribuir herramientas como AlphaFold. «Es bueno que lo hayan hecho, pero no deberíamos depender de ello», dice. «Necesitamos crear una infraestructura del sector público para poder hacerlo en la academia».